突变诱导耐药性数据库 (MdrDB)
关于 MdrDB
数据的全面性: MdrDB 旨从结构角度出发,构建一个蛋白质突变对小分子化合物结合亲和力影响的数据库。通过整合多种来源的突变诱导耐药性的相关信息。我们从 PDB 蛋白质结构数据库和 Uniprot 蛋白数据库获取蛋白质的结构和注释信息,从 PubChem 有机小分子生物活性数据库获取药物数据,从 GDSC 抗癌药物敏感性基因组学数据库和其他药物活性测试数据库获取药物活性变化等数据。
基于蛋白质结构: MdrDB 提供野生型蛋白质结构信息、突变型蛋白质结构信息、野生型蛋白质-配体复合物结构信息和突变型蛋白质-配体复合物结构信息,其可用于蛋白质突变研究和耐药性建模。
蛋白质突变的多样性: MdrDB 包含多种突变类型。除了单点突变外,还包含多种突变类型,如删除突变、插入突变、删除插入突变、多位点混合突变等复杂突变。
当前版本: 当前版本是 MdrDB v.1.0. 2022,包含 100537 个样本,由 240 种蛋白质(总共 5119 个 PDB 结构)、2503 个突变和 440 种药物组合而成。 其中,95971 个样本基于已知的PDB 结构,4566 个样本基于 AlphaFold2 预测结构。
更多信息请参阅教程。
| 数据集 | 样本数量 | 蛋自类型 | 结构化 | 突变类型 | ||
|---|---|---|---|---|---|---|
| 单点突变 | 多点突变 | 复杂突变 | ||||
| Platinum | 1,008 | 多种蛋白类型 | × | |||
| TKI | 144 | 激酶类型 | × | × | ||
| RET | 56 | 激酶类型 | × | × | ||
| AIMMS | 311 | 多种蛋白类型 | × | × | ||
| KinaseMD | 79 | 激酶类型 | × | × | ||
| MdrDB | 100,537 | 多种蛋白类型 | ||||
-
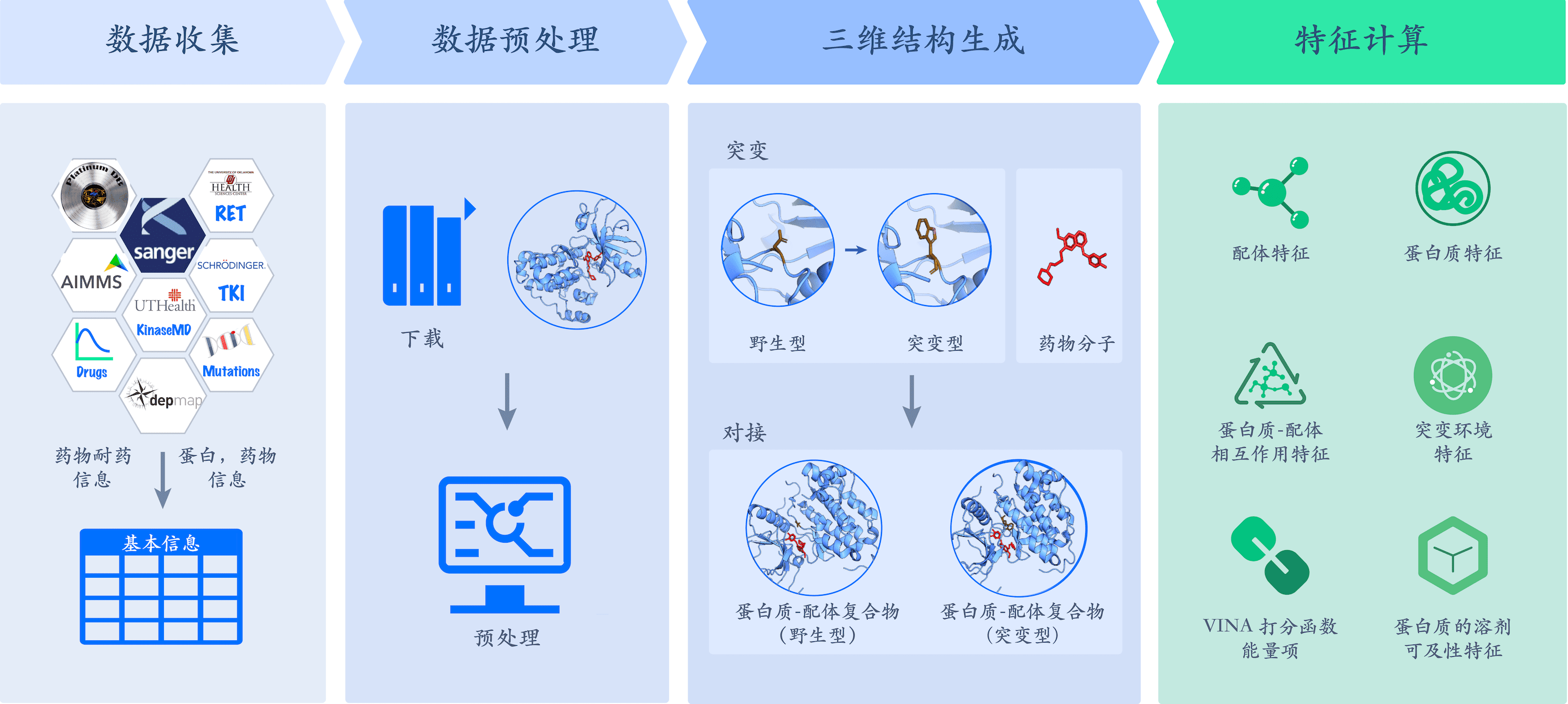
数据收集
-
数据预处理
蛋白质预处理: 首先为每个样本中的蛋白质找到对应的 Uniprot ID,并根据此 ID 在 PDB 数据库中查找和下载所有相应的可用结构(.pdb/.cif)和序列(.fasta)。然后,对于每个 PDB 结构,去除水分子和溶剂分子,并将蛋白质链和结合配体进行拆分。随后,通过检查 PDB 链中的突变位置信息以剔除无关的 PDB 结构。最后,我们对样本中蛋白质家族、超家族和结构域进行了查询和注释。
药物预处理: 首先对药物 SMILES 进行清理,去除所有的盐并中和电荷。然后,根据 SMILES生成药物 3D 结构并添加极性氢。最后,我们对药物的药理作用机制进行了查询和注释。
-
三维结构生成
基于可用的野生型蛋白质结构构建突变型蛋白质结构。对于简单的单点突变,突变型蛋白质结构从 Pymol Mutagenesis Wizard 模块产生的一系列突变残疾旋转异构体中选取能量最低的异构体获得。对于复杂突变,通过利用 AlphaFold2 和突变蛋白序列来预测突变体蛋白质结构。随后,将药物与野生型和突变型蛋白进行对接以获得蛋白质-配体复合物结构。
-
特征计算
总共计算了 146 个特征,可分为六种类型:18 个描述配体特性的特征,12 个描述野生型和突变蛋白差异的特征,21 个描述突变环境的特征,6 个描述蛋白质-配体相互作用的特征,59 个VINA 打分函数能量项和 30 个与配体和蛋白质的溶剂可及性相关特征。
统计数据
-
100537样本
-
240蛋白质
-
440药物
-
2503突变
